「プロジェクトHOPE (High-tech Omics-based Patient Evaluation)」(がんのマルチオミクス解析)
手術で取り出したがん組織や血液の細胞から遺伝子解析を行い、新しいがん診断・治療技術の開発につなげるプロジェクトを紹介しています。
プロジェクトHOPEの目標
プロジェクトHOPEは、2014年1月から開始された。第一次となる3年間の研究期間内に、静岡がんセンターで外科手術を受けるがん患者の臨床試料(手術時に摘除されたがん・周辺正常組織、血液)及び臨床情報を収集している。
プロジェクトの目標は、試料のマルチオミクス解析と臨床情報を統合解析し、がんの本態解明のため細胞のがん化(分裂・不死化・分化)や微小環境の変化(浸潤・転移・血管新生・免疫抑制)などが生じるメカニズムを明らかにすることにある。その過程で、腫瘍マーカーやバイオマーカーの開発がすすめられる。また、既存の抗がん剤の適応拡大さらには新規分子標的薬の標的探索がおこなわれる。実地医療の観点からは、患者ごとの臨床情報とオミクス情報が照合されることから、personalized medicine(個別化医療)をさらに発展させたindividualized medicine(個の医療)を実現し、ひいてはがん患者の全人的医療の実現を指向している。
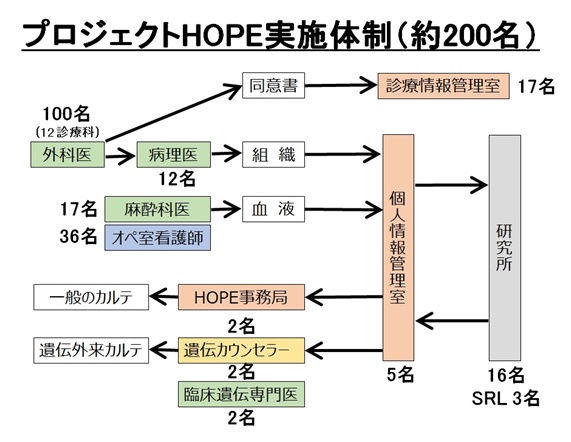
実施体制
プロジェクトHOPEの実施主体は静岡がんセンター病院及び研究所であり、職員約200名が参加している。株式会社エスアールエル(SRL)からは共同研究契約により、3名の技術人材が派遣されている。プロジェクトに対して国等からの経済的な支援はなく、独自の予算で実施している。
静岡がんセンターにおけるプロジェクトHOPE実施体制。
静岡がんセンターあげての取り組みで約200名の様々な職種が参加している。
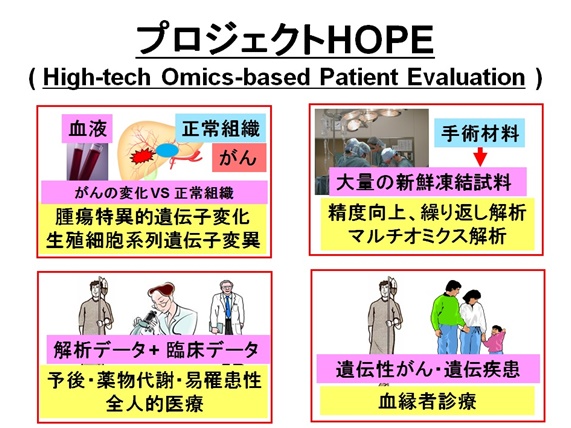
プロジェクトの特徴
プロジェクトHOPEの特徴と実験デザインを下記の図に示した。がん患者の手術時の摘除組織及び血液について、体細胞遺伝子変異及び生殖細胞系列遺伝子変異の両面から解析される。新鮮な手術摘除組織が十分量、凍結保存されるため、解析精度が確保でき、試薬等などが変更されたときにも繰り返し解析して検証できる。さらにマルチオミクス(ゲノミクス、トランスクリプトミクス、プロテオミクス、メタボロミクス)解析が可能となる。
個々の患者ごとに、解析データ(遺伝子構造変化と遺伝子発現変化)と臨床データが統合された臨床・マルチオミクス統合データベースが構築される。このデータベースは、将来の個の医療実現の基本となり、がんの易罹患性の評価、がん患者の予後予測のほか、特定薬剤の薬物代謝酵素遺伝子の遺伝子多型データなどから効果・副作用予測などにも役だつものである。
このプロジェクトでは、生殖細胞系列遺伝子変異が解析されることから、遺伝性がんに加えて、遺伝性疾患のリスク診断にも応用可能で、その結果は、本人のみでなく血縁者にも影響を与えることとなる。この分野は、いわゆるincidental findings(偶発的所見)として生命倫理学的な議論が始まっており、後に述べるようにプロジェクトHOPEのデータが、我が国では先鞭をつけ、全国的に注目を集めている。
患者試料及び倫理的対処について
静岡がんセンターで外科手術によりがん組織が摘出される患者は年間約3,000例に達する。その中で、試料提供に文書同意し、かつ解析に十分な量の組織が確保できる年間約1,000例から試料が採取されている。多くはステージIIまたはIIIの患者であり、早期がん(ステージI)はほとんど含まれない。したがって、対象となる患者の約半数が5年以内に再発や転移を起こすと予測される。
患者の臨床データは電子カルテ上に保管され、手術時の摘出組織試料(約0.5g)と血液はセットで保存される。組織については腫瘍組織と共に周辺の正常組織の双方が採取され、新鮮凍結組織と病理標本が保存される。血液は血球細胞と血漿とにそれぞれ分離され、凍結保存される。臓器別にみると、組織を採取しやすい大腸・直腸、肺、胃、頭頸部、乳腺、肝、肝転移、膵の順に多く、これらのがんが8割以上を占める。
臨床試料は、静岡がんセンターから持ち出すことはできない。患者から取得するインフォームドコンセントには共同研究先企業でも使用を可とすると記載されているが、その場合には共同研究契約を締結の上でセンター内のレンタルラボでの解析が許される。また、患者の臨床情報はすべてが開示されることはなく、必要性のあるもののみが共同研究を担当するセンター研究所の研究者にのみ開示される。
一方、静岡がんセンターでは生殖細胞系列の遺伝子解析により得られた偶発的所見を患者に開示している。開示される変異には、米国臨床遺伝学会で勧告された臨床上対応可能な28疾患56遺伝子以外に、最近、がん関連で報告のあったいくつかの遺伝子変異も含まれている。薬物代謝酵素の遺伝子多型や生活習慣病の発症リスクに有意に関わる遺伝子変異が見つかった場合には、治療法選択や生活習慣の改善指導に利用されている。遺伝カウンセラーは2名が配置されている。
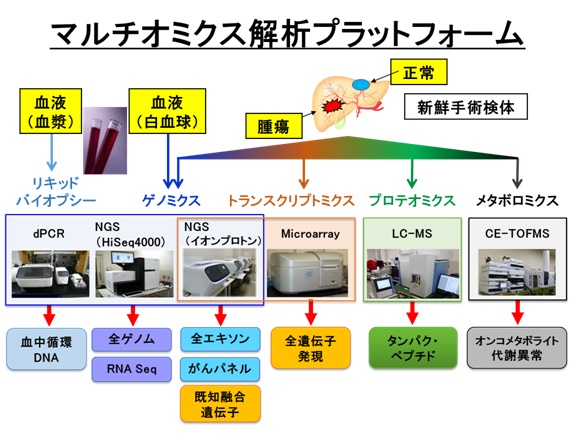
解析技術
遺伝子解析
Life TechnologiesのIon Proton system7台とIlluminaのMiSeqシステム2台がこのプロジェクトで稼働しており、それに加えてHiseq 4000の稼働準備が進められており、全ゲノムシーケンスも可能となる。
Ion Proton systemにおける全エキソン解析では、毎日5〜8例分のエキソンシーケンス(The Ion AmpliSeq Exome Kitを使用し、18,835遺伝子の変異を検出できる)を行っている。解析用の核酸ライブラリーの調製(前処理)に2〜3日を要する。シークエンスは1サンプルあたり半日で終了する。シークエンス結果が疑わしい場合には、サンガー法、パイロシークエンス法又はデジタルPCR法により検証し、精度を保持している。体細胞変異と生殖細胞系列変異の両方の変異を参照し、個々のサンプルについて、体細胞変異から生殖細胞系列変異を差し引いて体細胞に固有の変異を特定している。
SNVの解析に加え、独自に開発した検出系(HOPE Fusion Panel)により490の既知がん融合遺伝子の有無も同時に調べている。
解析を終えた全症例について、がん関連遺伝子の種類別に分類し、変異数順で見てみると、上位の遺伝子は、TP53(40%で、米国のデータベースとほぼ同じ)、APC(大腸がんで多く発現)、CDC27、KRAS、SYNE1であった。
また、がんの発生した臓器別で分類すると、TP53のように大部分のがんで共通して変異が見られる遺伝子と、APCのように特定のがんのみで変異が見られる遺伝子とが存在した。前出の腫瘍特異的SNV数も、また、がんの種類別の遺伝子変異の特性も、多数の日本人がん患者を対象としたデータとしては、プロジェクトHOPEの研究デザインによって初めて明らかにされた所見と言える。
遺伝子発現解析
Agilent TechnologiesのDNAマイクロアレイを使用している。mRNAの品質管理はAgilent TechnologiesのRIN(RNA Integrity Number)を使用し、RIN値が6.0以上のものを分析のい用いている。
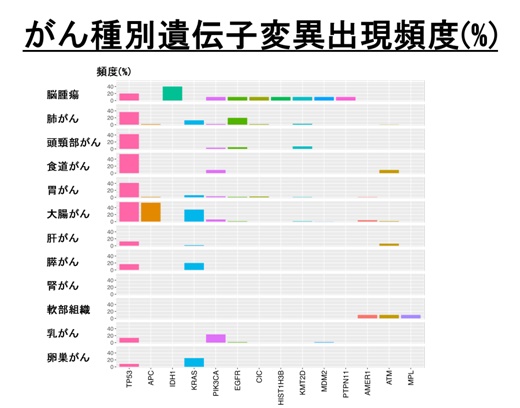
がんの種類別にみたがん関連遺伝子変異の出現頻度
プロテオミクス
がん細胞が特異的に産生するタンパク質を同定するための質量分析による構造解析研究、さらにその細胞内での機能、血液中での分子存在型、濃度、腫瘍特異性等の解析研究を行い、腫瘍マーカーあるいはバイオマーカーとして有用であるタンパク質の測定系の開発を目指している。
メタボロミクス
がん細胞においては、遺伝子の変異や発現変化に伴い、細胞内の代謝が変化する。この変化をとらえることにより、新しい治療標的やバイオマーカーの開発を目指している。
免疫チェックポイント阻害剤のバイオマーカー等
プロジェクトHOPEでは、以下について検証を行う予定である。
1) PD-L1を産生する腫瘍での免疫チェックポイント阻害剤の薬効
2) 遺伝子変異の多い(hyper-mutation)腫瘍での阻害剤の効果の検証
3) PD-L1以外の候補分子を発現する腫瘍での検証
免疫チェックポイント阻害剤(PD-1抗体医薬、ニボルブマブ)の薬効を予測するバイオマーカーの検索のために、免疫関連の100遺伝子の発現解析を行っている。
SNVが多数存在するがん(hyper mutation load)は、免疫チェックポイント阻害剤(PD-1、PD-L1抗体医薬)の奏功が期待できると報告されている。変異数が新しいバイオマーカーになるかもしれない。治療用のキメラ及びヒト化抗体を作製できる体制を整えている。
薬効評価系として改良型NOGマウスを用いたゼノグラフトモデルを構築中である。このマウスは、免疫チェックポイント阻害剤の薬効評価に使用できる。
免疫細胞療法をさらに多くのがん患者に適用するために、次世代シーケンサーを用いて、遺伝子変異ペプチドを同定している。HLA ペプチドの結合予測とin vitro、in vivo評価でしぼりこみ、ペプチドワクチンの開発も計画中である。
今後の展望と課題
プロジェクトHOPEの目標は、患者個々人向けの(1)がんの個別化医療の推進、(2)未病医学の実践(予防的、発症前治療)、(3)医療スタッフ・研究者の学習、(4)がんの診断薬・治療薬の研究と開発の4点であり、それぞれの展望と課題は以下の通りである。
(1) がんの個別化医療の推進
プロジェクトHOPEは、遺伝的背景に基づき個々の患者に適した治療を目指している医療スタッフと研究者達が意見を出し合い、病院ベースで立ち上げられたプロジェクトであり、このような背景は、がん患者の個別化医療を強く推進している。例えば本プロジェクトに登録された患者のほとんどはステージⅡまたはⅢの患者であり、これら患者の約半数は今後がんを再発するリスクを有している。プロジェクトHOPEでは、個々の患者のがんの性質、特にがんのドライバー遺伝子やがんの微小環境の調節因子に関する情報が得られるため、がんが再発した場合に、個々のがんの特性に見合った分子標的薬の選択を確実かつ迅速に行うことができる。
また、マルチオミクス解析に基づく医療は、医学の世界においても始まったばかりで、その方向性については不透明な部分もあるが、本領域における技術の進歩は著しい。プロジェクトHOPEに関して言えば、今後、全ゲノムシークエンスやエピジェネティック解析による試料の再調査が必要になるであろう。プロジェクトHOPEでは、豊富な量のがん及び正常組織試料が確保されているので、新規方法論や技術の進展に応じて、繰り返しサンプルの利用、解析が可能である。
(2) 未病医学の実践(予防的、発症前治療)
プロジェクトHOPEは、「予防的・発症前治療」又は「未病医学」の実践の機会を提供する。血液細胞を用いた生殖細胞系列の全キソン解析結果より得られる患者体質に関する遺伝情報より、遺伝性がんのみならず非がん性遺伝性疾患の発症リスクを予見できるため、予防的・発症前治療が可能になる。このことは、患者のみならず血縁者にとっても有用であり、適切な医学的介入により、罹患率や死亡率を低下できることは注目すべきである。
(3) 医療スタッフ・研究者の学習
プロジェクトHOPEは、医療スタッフと研究者にマルチオミクス解析について学ぶ機会を提供する。静岡がんセンターの医療スタッフは、患者のがんと体質に関する遺伝的変化について学んだ後に、患者を診療することになる。このことは、がん治療の改良に繋がるだけでなく、医療スタッフがゲノム医療を習得することの手助けとなり、遺伝的変化と臨床症状とが如何に関連しているかについて、より詳細に学ぶようになる。その結果、遺伝子解析に基づく臨床検査が、より広く認知され、社会的なコンセンサスが形成されることに繋がる。
(4) がんの診断薬・治療薬の研究と開発
プロジェクトHOPEの完遂により、がん医学において重要な研究開発のシーズが創出される。がん組織の解析で見つかった新規の遺伝的変化が、新規分子標的薬剤、バイオマーカー、及び腫瘍マーカー等の開発に繋がることが期待される。
公益財団法人ヒューマンサイエンス振興財団(http://www.jhsf.or.jp/)創薬基
盤研究推進事業 平成27年度創薬資源調査報告書「医療ビックデータの活用並び
にバイオマーカー実用化の最新動向 -創薬並びに個別化医療や予防医療への可
能性を探る-」 72頁~83頁より、一部改変して掲載しております。
報告書の全文は、下記ホームページにてご確認ください。
http://www.jhsf.or.jp/paper/report/report_201504.pdf
主な研究成果 論文
| ▶ | Nagashima T, Yamaguchi K, Urakami K, Shimoda Y, Ohnami S, Ohshima K, Tanabe T, Naruoka A, Kamada F, Serizawa M, Hatakeyama K, Matsumura K, Ohnami S, Maruyama K, Mochizuki T, Kusuhara M, Shiomi A, Ohde Y, Terashima M, Uesaka K, Onitsuka T, Nishimura S, Hirashima Y, Hayashi N, Kiyohara Y, Tsubosa Y, Katagiri H, Niwakawa M, Takahashi K, Kashiwagi H, Nakagawa M, Ishida Y, Sugino T, Takahashi M, Akiyama Y. Japanese version of The Cancer Genome Atlas, JCGA, established using fresh frozen tumors obtained from 5143 cancer patients. Cancer Sci. 2020;111(2):687-699. |
| ▶ | 長嶋剛史, 山口建, 浦上研一, 下田勇治, 大浪澄子, 大島啓一, 田邉智絵, 成岡茜, 鎌田福美, 芹澤昌邦, 畠山慶一, 松村研哉, 大浪俊平, 丸山宏二, 望月徹, 楠原正俊, 塩見明生, 大出泰久, 寺島雅典, 上坂克彦, 鬼塚哲郎, 西村誠一郎, 平嶋泰之, 林央周, 清原祥夫, 坪佐恭宏, 片桐浩久, 庭川要, 髙橋かおる, 柏木広哉, 中川雅裕, 石田裕二, 杉野隆, 高橋満, 秋山靖人. 日本版がんゲノムアトラス(JCGA, Japanese version of The Cancer Genome Atlas)の構築 -日本人がん患者5143人から得た新鮮凍結腫瘍組織の解析-. ※「Japanese version of The Cancer Genome Atlas, JCGA, established using fresh frozen tumors obtained from 5143 cancer patients」をもとに、一部新たなデータを追加し、補完的な図表を本文中に配置した日本語版です。 |
| ▶ | Hatakeyama K, Nagashima T, Ohshima K, Ohnami S, Ohnami S, Shimoda Y, Serizawa M, Maruyama K, Naruoka A, Akiyama Y, Urakami K, Kusuhara M, Mochizuki T, Yamaguchi K. Mutational burden and signatures in 4000 Japanese cancers provide insights into tumorigenesis and response to therapy. Cancer Sci. 2019;110(8):2620-2628. |
| ▶ | Hatakeyama K, Ohshima K, Nagashima T, Ohnami S, Ohnami S, Serizawa M, Shimoda Y, Maruyama K, Akiyama Y, Urakami K, Kusuhara M, Mochizuki T, Yamaguchi K. Molecular profiling and sequential somatic mutation shift in hypermutator tumours harbouring POLE mutations. Sci Rep. 2018;8(1):8700. |
| ▶ | Ohnami S, Nagashima T, Urakami K., Shimoda Y, Kamada F, Saito J, Naruoka A, Serizawa M, Masuda Y, Ohnami S, Kusuhara M, Yamaguchi K. Whole exome sequencing detects variants of genes that mediate response to anticancer drugs. J. Toxicol. Sci. 2017;42(2):137-144. |
| ▶ | Shimoda Y, Nagashima T, Urakami K, Tanabe T, Saito J, Naruoka A, Serizawa M, Mochizuki T, Ohshima K, Ohnami S, Ohnami S, Kusuhara M, Yamaguchi K. Integrated next-generation sequencing analysis of whole exome and 409 can-cer-related genes., Biomed Res. 2016;37(6): 367-379. |
| ▶ | Nagashima T, Shimoda Y, Tanabe T, Naruoka A, Saito J, Serizawa M, Ohshima K, Urakami K, Ohnami S, Ohnami S, Mochizuki T, Kusuhara M, Yamaguchi K. Optimizing an ion semiconductor sequencing data analysis method to identify somatic mutations in the genomes of cancer cells in clinical tissue samples. Biomed Res. 2016;37(6):359-366. |
| ▶ | Yamaguchi K, Urakami K, Nagashima T, Shimoda Y, Ohnami S, Ohnami S, Ohshima K, Mochizuki T, Hatakeyama K, Serizawa M, Akiyama Y, Maruyama K, Katagiri H, Ishida Y, Takahashi K, Nishimura S, Terashima M, Kawamura T, Kinugasa Y, Yamakawa Y, Onitsuka T, Ohde Y, Sugino T, Ito I, Matsubayashi H, Horiuchi Y, Mizuguchi M, Yamazaki M, Inoue K, Wakamatsu K, Sugiyama M, Uesaka K, Kusuhara M. Prevalence of low-penetrant germline TP53 D49H mutation in Japanese cancer patients. Biomed Res. 2016;37(4):259-264. |
| ▶ | Urakami K, Shimoda Y, Ohshima K, Nagashima T, Serizawa M, Tanabe T, Saito J, Usui T, Watanabe Y, Naruoka A, Ohnami S, Ohnami S, Mochizuki T, Kusuhara M, Yamaguchi K. Next generation sequencing approach for detecting 491 fusion genes from human cancer. Biomed Res. 2016;37(1):51-62. |
| ▶ | Yamaguchi K, Urakami K, Ohsima K, Mochizuki T, Akiyama Y, Uesaka K, Nakajima T, Takahashi M, Tamai S, and Kusuhara M. Implementation of individualized medicine for cancer patients by multiomicsbased analyses—the Project HOPE—. Biomedical Research. 2014;35(6):407-412. |